Desde a conclusão do primeiro grande Projeto Genoma, em 2003, houve um grande avanço no desenvolvimento de novas tecnologias de e novas plataformas de sequenciamento de DNA.
Até então, a metodologia mais amplamente utilizada era o método de Sanger, automatizado em 1998 pela Applied Biosystems (ABI). Resumidamente, esta metodologia consistia em uma reação de PCR (Polymerase Chain Reaction) utilizando nucleotídeos marcados (dideoxinucleotídeos), e não marcados. Conforme a reação acontecia, algumas moléculas de DNA incorporavam estes nucleotídeos marcados, podendo ser identificados posteriormente pela emissão de fluorescência, mas que na sua origem fazia uso de radioatividade.
Sequenciamento de Nova Geração – NGS
O genoma humano demorou aproximada 10 anos para ser sequenciado. Na última década, passamos por uma revolução tecnológica que nos permite sequenciar o genoma humano completo em menos de uma semana. Estas tecnologias tiveram seus desenvolvimentos iniciados na década de 90, e culminaram nas plataformas de sequenciamento de nova geração (NGS) que conhecemos e que ainda estamos por conhecer.
O pirosequenciamento (454 Roche), foi a primeira plataforma de sequenciamento de nova geração disponibilizada para o mercado em meados de 2005. Esta tecnologia causou bastante impacto, pois dispensava a necessidade de clonagem prévia de fragmentos e proporcionava o sequenciamento de milhares de sequenciamentos de DNA e amostras em paralelo, onde todo o DNA presente na amostra era diretamente clivado por shotgun, e um PCR em emulsão de moléculas únicas era realizado. Esta metodologia baseia-se no princípio de detecção de luz a partir de um pirofosfato liberado na reação, no momento em que cada nucleotídeo específico é incorporado na molécula de DNA sendo sequenciada.
De 2005 para frente, outras metodologias surgiram baseadas no princípio de detecção da fluorescência de terminadores reversíveis. O sequenciamento por síntese é realizado em paralelo para um grande número de amostras de DNA imobilizadas em uma superfície, onde a fluorescência de cada nucleotídeo é detectada por uma câmera CCD. Esta abordagem de sequenciamento adotado pela 454, Illumina, SOLiD permitiu uma grande combinação de informações quantitativas e qualitativas possibilitando um enorme avanço nas análises genômicas que eram antes tecnicamente impossíveis, ou muito caras.
Mais recentemente, uma nova geração de sequenciadores encontra-se em desenvolvimento, baseados na ausência de amplificação prévia da amostra e detecção de uma única molécula. Entre estas tecnologias destacam-se principalmente: a Heliscope, o SMRT da Pacific-Bioscience e o Oxford Nanopore, que utilizam desde polimerases modificadas imobilizadas em uma superfície até nanoporos que detectam variações de corrente elétrica, ou ainda, a liberação de íons resultantes do processo de polimerização da molécula nascente, como na tecnologia Ion.
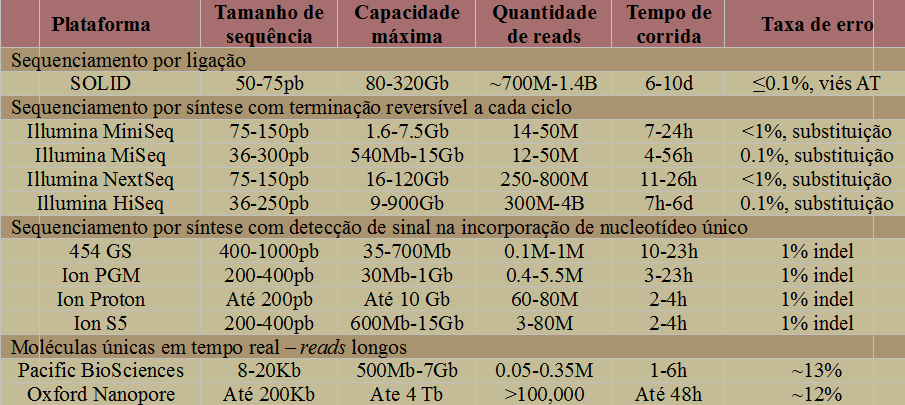
Todas estas plataformas desenvolvidas hoje fazem já parte das rotinas de laboratório, sendo que, cada uma possui suas vantagens e especificidades. Algumas comparações podem ser feitas (tabela abaixo), contudo, este é um campo em constante desenvolvimento e aprimoramento, possibilitando cada vez mais a geração de uma maior quantidade de sequências, maior multiplexagem de amostras, com maior tamanho de fragmentos, melhor qualidade e menor custo por base sequenciada.
doi:10.1038/nrg.2016.49
Algumas referências para saber mais sobre as metodologias de sequenciamento:
- Applied Biosystems. 2011. SOLiD System accuracy with the Exact Call Chemistry module, 1–8.
- Bayley H. 2006. Sequencing single molecules of DNA. Current Opinion in Chemical Biology 10, 628–637.
- Bowers J, Mitchell J, Beer E, et al. 2010. Virtual Terminator nucleotides for next generation DNA sequencing. Nature Methods 6, 593–595.
- Braslavsky I, Braslavsky I, Hebert B, Hebert B, Kartalov E, Kartalov E, Quake SR, Quake SR. 2003. Sequence information can be obtained from single DNA molecules. Proceedings of the National Academy of Sciences of the United States of America 100, 3960–4.
- Clarke J, Wu H, Jayasinghe L, Patel A, Reid S, Bayley H. 2009. Continuous base identification for single-molecule nanopore DNA sequencing. Nature nanotechnology 4, 265–270.
- Eid J, Fehr A, Gray J, et al. 2009. Real-time DNA sequencing from single polymerase molecules. Science (New York, N.Y.) 323, 133–138.
- Mardis ER. 2013. Next-Generation Sequencing Platforms. Annual Review of Analytical Chemistry 6, 287–303.
- McKernan KJ, Peckham HE, Costa GL, et al. 2009. Sequence and structural variation in a human genome uncovered by short-read, massively parallel ligation sequencing using two-base encoding. Genome Research 19, 1527–1541.
- Ozsolak F, Platt AR, Jones DR, Reifenberger JG, Sass LE, McInerney P, Thompson JF, Bowers J, Jarosz M, Milos PM. 2009. Direct RNA sequencing. Nature 461, 814–818.

