O sequenciamento de nova geração vem ganhando um papel fundamental na investigação de dados biológicos e diferentes tecnologias têm melhorado o custo e desempenho das análises desses dados. A leitura feita no sequenciador pode ser feita de duas formas mais comuns: single-end e paired-end.
Atualmente, a identificação de patógenos em indústrias e produtos alimentícios ou a caracterização de comunidades microbiológicas em diferentes contextos já é realizada através do sequenciamento de nova geração de marcadores genéticos, como o gene da subunidade ribossomal 16S para bactérias, a região espaçadora ITS para fungos, a região codificadora da proteína citocromo B (CYTB) para animais ou a região intergênica psbA-trnH para plantas.
Além disso, com o barateamento das plataformas de sequenciamento e de seus insumos, o sequenciamento completo do genoma desses organismos também têm se mostrado uma alternativa viável para identificar espécies que apresentam um desafio à classificação taxonômica com marcadores clássicos, como Escherichia coli e Salmonella spp.
O sequenciamento inicia-se com a fragmentação do DNA, um processo realizado de maneira aleatória e que gera diversos pequenos fragmentos.
A leitura desses fragmentos pode ser feita de diferentes formas pelo sequenciador, sendo as duas mais comuns single-end e paired-end. É importante destacar que ao fazer a leitura desses fragmentos, independente do tipo de biblioteca, o sequenciador gera um read.
O que é single-end?
No sequenciamento de uma biblioteca preparada com a tecnologia single-end, um adaptador é adicionado em apenas uma extremidade do fragmento construído.
Esse adaptador é uma sequência de nucleotídeos previamente conhecida e que possibilita a acoplagem do mesmo ao sequenciador. Além disso, também é adicionado um primer que vai permitir o sequenciamento em um único sentido.
Essa abordagem garante a geração de uma quantidade grande de reads com tamanhos um pouco maiores e de forma barata que pode ser aplicada em estudos de CHIP-Seq ou até mesmo estudos mais simples de RNA-Seq.
O que é paired-end?
Na tecnologia paired-end, por outro lado, o fragmento a ser sequenciado possui um adaptador em cada ponta e dois primers vão permitir que o sequenciamento comece em um sentido, continue até uma determinada posição e, ao finalizar, a próxima rodada começará com o sequenciamento na ponta contrária, também avançando até o comprimento estimado.
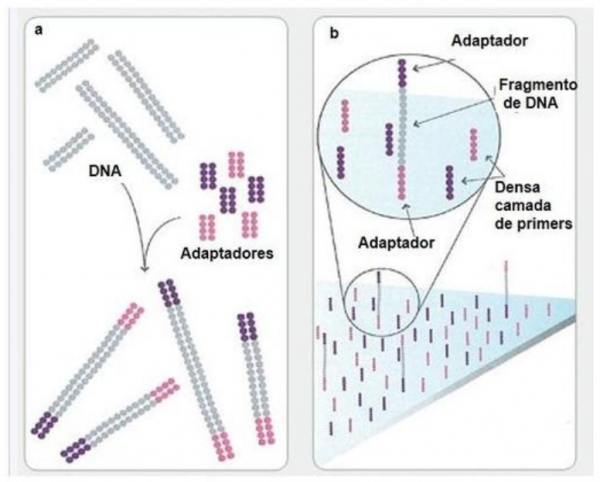
Fonte: CARVALHO, M.C.C.G & SILVA, C.G. (2010). Next generation DNA sequencing and its applications in plant genomics. Cienc. Rural 40 (3), 2010.
Essa simples abordagem de realizar a leitura de um fragmento pelas duas extremidades permite que exista uma confiança muito maior ao inferir a estrutura de uma determinada posição genômica depois que o alinhamento a uma sequência de referência tenha sido feita.
Essa informação pareada consegue melhorar significativamente a determinação de estruturas gênicas, facilitar a identificação de eventos de reorganização cromossômica, aprimorar a estimativa de expressões gênicas (mesmo a nível de transcritos alternativos) e até aperfeiçoar o processo de montagem de genomas que possuem níveis altos de sequências repetitivas.
Entretanto, sequenciamentos paired-end requerem um preparo de biblioteca mais trabalhoso e necessitam de mais insumos e tempo de sequenciamento para atingirem um rendimento equivalente ao single-end, o que torna essa tecnologia mais onerosa.
Apesar disso, o sequenciamento paired-end já tem sua popularidade consolidada e é amplamente utilizado pela comunidade científica em estudos como diagnóstico de doenças genéticas, classificação de novas espécies, identificação de patógenos, entre outros.
Como a Neoprospecta aplica essa tecnologia
Na Neoprospecta tanto as tecnologias single-end quanto paired-end são empregadas no sequenciamento.
Para análises de diversidade bacteriana através de Diagnóstico Molecular Digital (DMD) com o marcador 16S, por exemplo, os pipelines da Neoprospecta utilizam dados paired-end para garantir uma caracterização adequada da comunidade bacteriana analisada.
Também são utilizados dados paired-end em análises de sequenciamento de genoma completo, onde o emprego dessa tecnologia associada a uma cobertura elevada é imprescindível para identificar até mesmo grupos complexos, como sorotipos de Salmonella spp. ou, ainda, espécies de bactérias ou fungos que possuem relações taxonômicas intrincadas, como as bactérias da família Enterobacteriaceae, que possui integrantes com potencial patogênico.
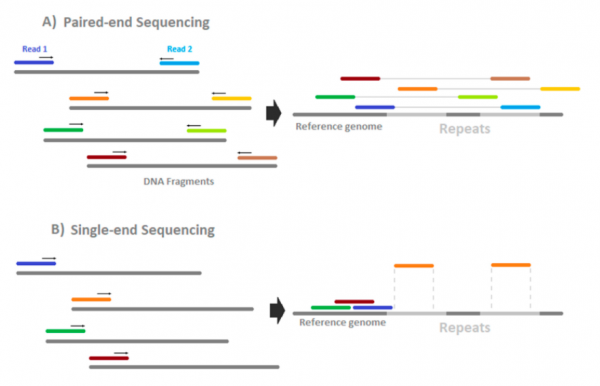
GREHL, C. et al (2018).How to Design a Whole-Genome Bisulfite Sequencing Experiment. Epigenomes 2(4), 21, 2018.
Tanto as tecnologias single-end quanto paired-end continuam sendo muito utilizadas e aceitas pela comunidade científica, o que de fato varia é sua finalidade, tempo disponível para processamento e complexidade do objetivo para qual serão aplicadas.
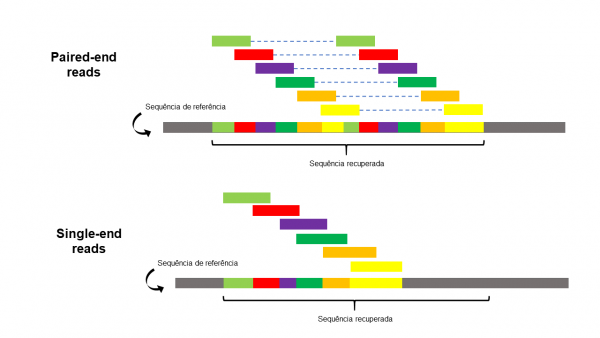
Fonte: Autor
Referências
https://systemsbiology.columbia.edu/genome-sequencing-defining-your-experiment
https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-020-3484-z
Autor
Igor Paim
Graduado em Biotecnologia com ênfase em Bioinformática pela Universidade Federal do Rio Grande do Sul e Bioinformata na Neoprospecta.

