Os bacteriófagos, ou fagos, são vírus capazes de infectar bactérias. Eles possuem diferentes aplicações na área de alimentos e podemos citar o controle de bactérias patogênicas e deteriorantes como exemplo. A identificação e caracterização de bacteriófagos é uma das análises oferecidas pela Neoprospecta, ela utiliza como metodologia o Sequenciamento do Genoma Completo, seguido por análises de bioinformática. O objetivo deste NeoScience é explicar como é realizada a análise de bacteriófagos na Neoprospecta.
Primeiramente, o que são bacteriófagos?
Os bacteriófagos, também conhecidos como fagos, são vírus capazes de infectar bactérias. Estes vírus não se multiplicam de forma independente, eles necessitam de um hospedeiro. Por este motivo, são normalmente encontrados em maior abundância em locais habitados por seus hospedeiros.
Os bacteriófagos são vírus bacterianos naturais, presentes em diversos locais, como água, solo e ar. Os fagos podem se diferenciar pelos seus ciclos biológicos, sendo assim, são classificados em líticos ou lisogênicos. Os líticos possuem alta taxa de replicação, provocando a lise da célula bacteriana. Já os lisogênicos tem seu material genético integrado ao da bactéria, havendo um silenciamento da expressão dos genes do fago.
Os bacteriófagos podem ser aplicados na área de alimentos de diferentes formas, sendo uma delas a utilização dos mesmos como forma de controle de bactérias patogênicas e deteriorantes.
Identificação e caracterização de bacteriófagos
A identificação e/ou caracterização de bacteriófagos utiliza como metodologia o Sequenciamento do Genoma Completo, do inglês Whole Genome Sequencing (WGS), seguido por uma acurada análise de bioinformática.
Esta análise pode ser realizada tanto apenas para identificação quanto para a caracterização de bacteriófagos. A diferença entre os dois está na etapa da análise de bioinformática.
Esta metodologia também é utilizada para outras aplicações, como a análise de Sorotipagem Molecular, que permite a identificação dos sorotipos de Salmonella e sua comparação com genomas de referência.
Sequenciamento completo do Genoma
A análise de Sequenciamento do Genoma Completo é a melhor forma de identificação de microrganismos. Para a sua realização, no caso de bacteriófagos, é necessário que as amostras sejam entregues no formato de DNA circularizado. Caso contrário, haverão perdas nas pontas do genoma, impossibilitando o sequenciamento completo. O DNA pode ser entregue pelo cliente ou ser extraído no próprio laboratório.
Assim que a amostra chega ao laboratório, inicia-se a etapa de preparo do DNA. Assim, o mesmo poderá entrar no equipamento MiSeq, estando apto para leitura, ou seja, para identificação dos pares de base.
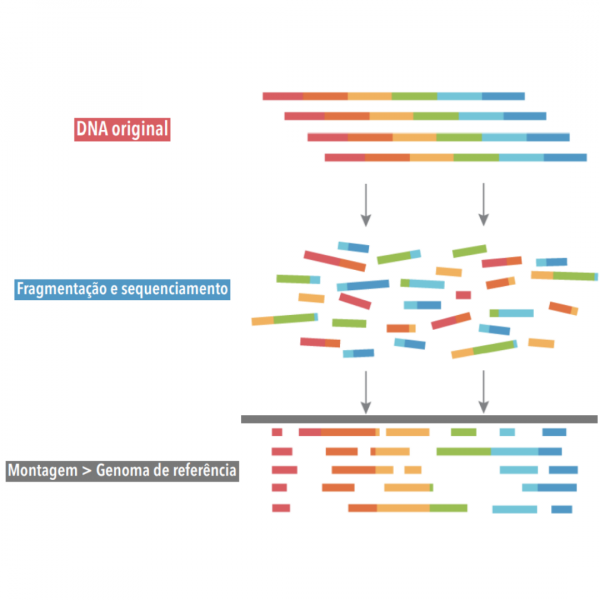
O DNA é clivado via shotgun em pequenos fragmentos, de cerca de 400-500 pares de base. Estes fragmentos são então ligados à adaptadores para o sequenciamento de DNA em Plataforma Illumina. Após o sequenciamento, são realizadas análises de bioinformática para montagem da sequência completa do genoma.
Análises de bioinformática
Com os fragmentos sequenciados, as análises de bioinformática são utilizadas para a montagem da sequência completa do genoma. Além disso, as sequências são comparadas com bancos de dados de referência. Estes bancos reúnem milhares de sequências de DNA de organismos conhecidos que foram classificados por pesquisadores do mundo inteiro. Assim, um genoma até então desconhecido pode ser comparado com diversos genomas presentes em um banco de dados, e por similaridade entre os genomas, utilizam-se ferramentas de bioinformática para identificação do genoma recém montado.
Após a etapa de sequenciamento, programas de bioinformática são utilizados para avaliação da Qualidade Phred, que indica a chance de cada base ter sido ou não corretamente identificada durante o sequenciamento. Sequências com valores de Qualidade Phred baixos são eliminadas.
Um ou mais programas podem ser utilizados para a montagem do genoma, eles transformam pequenas sequências em sequências maiores, conhecidas como Scaffolds. Adicionalmente, podem ser utilizados programas para melhorar a qualidade de uma montagem. No pipeline utilizado pela Neoprospecta, existe um programa que atua como uma espécie de mediador, ou seja, ele une os melhores resultados produzidos por dois montadores de genoma, originando uma terceira montagem, mais refinada. E é a partir desta montagem que os resultados são entregues aos clientes.
Sendo assim, a etapa de montagem basicamente parte do DNA circularizado, seguindo para a fragmentação, sendo esta realizada em laboratório. Nesta etapa, algo que possui milhares de pares de base se transforma em fragmentos de centenas de pares de bases (reads). Estes reads possuem tamanhos semelhantes e são ordenados pelos programas de montagem de genoma anteriormente citados. Após isso, a sobreposição das bases da sequência de nucleotídeos gera uma sequência conhecida como Contig, e a sobreposição desses Contigs gera um SuperContig ou Scaffold, que seria um refinamento de um ou mais Contigs.
Terminada estas etapas, possuímos alguns Scaffolds, compostos por milhares ou milhões de pares de bases e precisamos identificar as regiões que codificam alguma proteína. Um programa é então utilizado para realizar a predição das regiões que codificam ou não uma proteína. Este programa permite identificar onde inicia e onde termina um gene. Após isso, são utilizados genomas para identificação da espécie, podendo utilizar tanto bancos próprios quanto públicos. Ou seja, a partir do conteúdo de Scaffolds que passaram pelos parâmetros de qualidade, é possível a identificação da espécie.
Os bacteriófagos são frequentemente associados às espécies de bactérias, os pipelines de anotação permitem a identificação do hospedeiro a qual o bacteriófago pertence. Além disso, é possível comparar genomas montados com os nossos pipelines com genomas de bacteriófagos publicados na literatura, verificando orientação do genoma, sintenia, bem como utilizar proteínas específicas de determinados bacteriófagos para identificações filogenéticas, obtendo estudos evolutivos dos bacteriófagos.
Quer saber mais sobre a análise de identificação e/ou caracterização de bacteriófagos da Neoprospecta?
Clique aqui e fale com nossos especialistas!

