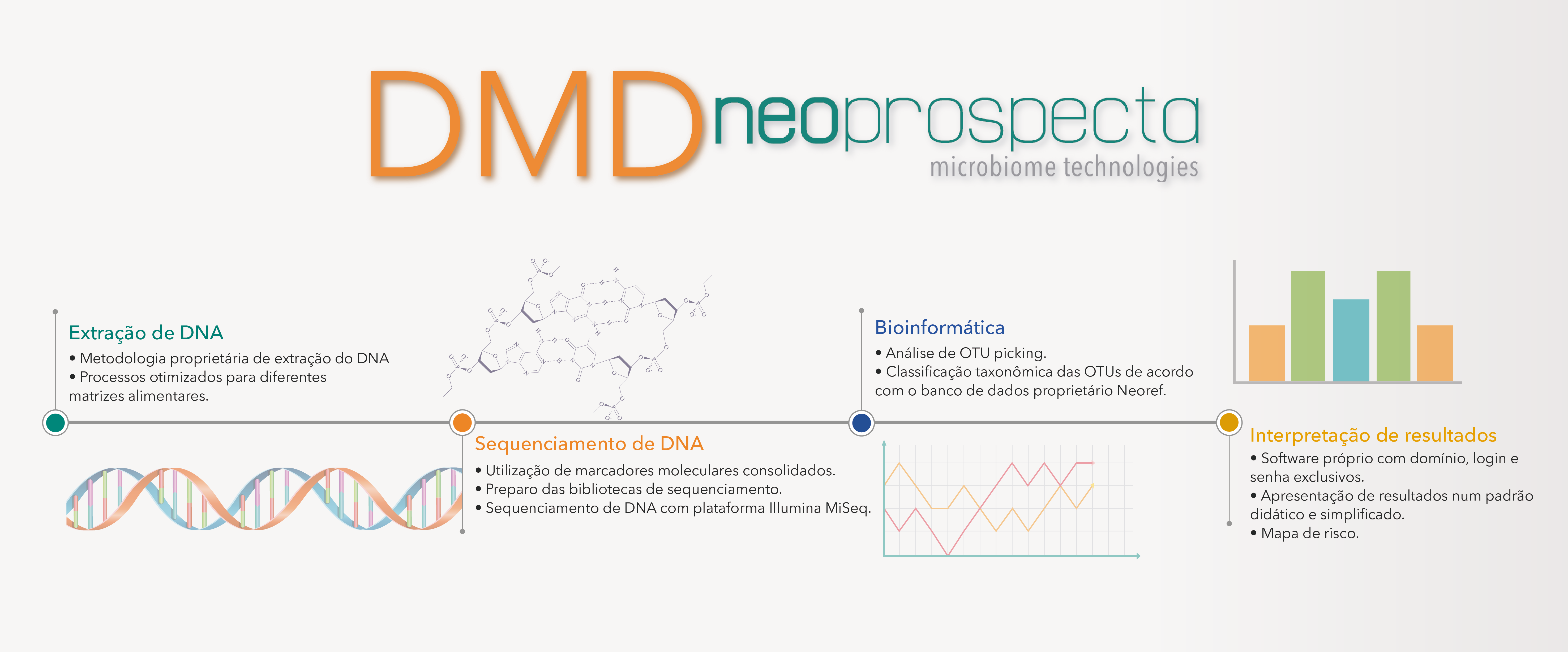
A metodologia citada nesse artigo, tem por objetivo a identificação específica de microrganismos utilizando técnicas de sequenciamento de DNA em larga escala (high-throughput sequencing) e de nova geração (NGS).
A qualidade dos dados gerados e sua alta sensibilidade, permitem a identificação acurada das espécies citadas, já demonstrando um resultado final sem a necessidade de testes confirmatórios adicionais.
Informações sobre a metodologia
A abordagem DMD Food Plex1, consiste em um painel de genes marcadores, desenvolvidos especificamente para a detecção acurada, ao nível de espécie, de Salmonella enterica, Listeria monocytogenes, Vibrio parahaemolyticus, Staphylococcus aureus, Bacillus* e Escherichia coli, além de identificar de forma mais ampla as bactérias mesófilas aeróbias presentes na amostra.
Essa abordagem viabiliza o monitoramento da qualidade dos alimentos, identificando a presença dos microrganismos contaminantes compreendidos no painel Plex 1, utilizando apenas um ensaio para a obtenção dos resultados.
Características da metodologia
- Painel de genes selecionados para identificação específica de bactérias por sequenciamento de DNA;
- Alta sensibilidade para detecção de bactérias patogênicas de interesse em alimentos;
- Resultados confirmados por metodologias de microbiologia convencional;
- Maior resolução sobre a identidade dos microrganismos presentes em uma amostra;
- Metodologia simples e rápida para confirmação dos microrganismos.
Quer conhecer mais sobre DMD Food?
O artigo abaixo apresenta os resultados obtidos com o método DMD Food Plex1 e a detecção de microrganismos de interesse para a segurança dos alimentos.
Clique na imagem abaixo e acesse o artigo completo com toda a metodologia detalhada.